背景
HCV NS3/4A 蛋白酶 (Protease,PR) 是由NS3丝氨酸蛋白与辅助因子NS4A 蛋白以非共价键结合而成的二聚体。NS3 丝氨酸PR 是HCV非结构蛋白加工成熟过程的关键酶,与三磷酸核苷酸酶、RNA 解螺旋酶结合,构成HCV 复制所必需的非结构蛋白NS3。NS3 属于胰PR 超家族,拥有PR 和RNA 解螺旋酶双重活性,位于由HCV RNA 编码的前体多聚蛋白的开放阅读框 (Open reading frame,ORF) 中的非结构蛋白区中。另外,NS3/4A PR 能水解TRIF 和MAVS 两种人类免疫蛋白,从而干扰人体对HCV 的先天性免疫应答。NS3/4A PR 还可通过调控IFN-3 的表达水平,破坏细胞内免疫通路,诱导宿主产生免疫逃逸。
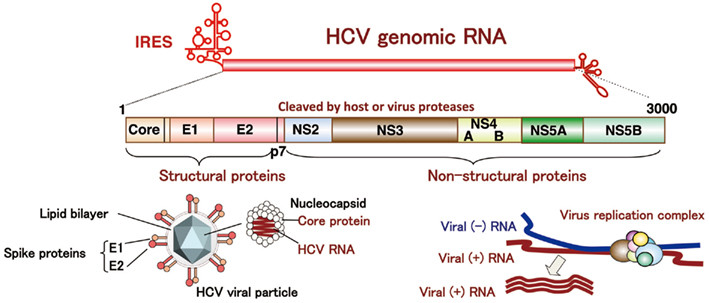
Faldaprevir 类似物分子 (Faldaprevir analogue molecule ,FAM)作为一种HCVNS3/4A PR Inhibitor,能有效抑制NS3/4A PR 活性。阻断HCV 的复制、翻译和翻译后多聚蛋白的加工成熟,还可使IFN 更好地发挥疗效。
目的
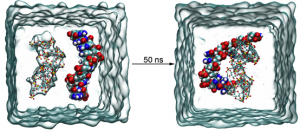
用分子动力学 (Molecular dynamics,MD) 模拟方法对HCV NS3/4A 蛋白酶与Faldaprevir 类似物的分子识别机制及其复合物运动模式进行了研究,为后续的结构改造提供结构及理论支撑。
方法
分子动力学模拟(Amber)
能量分解(MM/GBSA)
主要研究结果
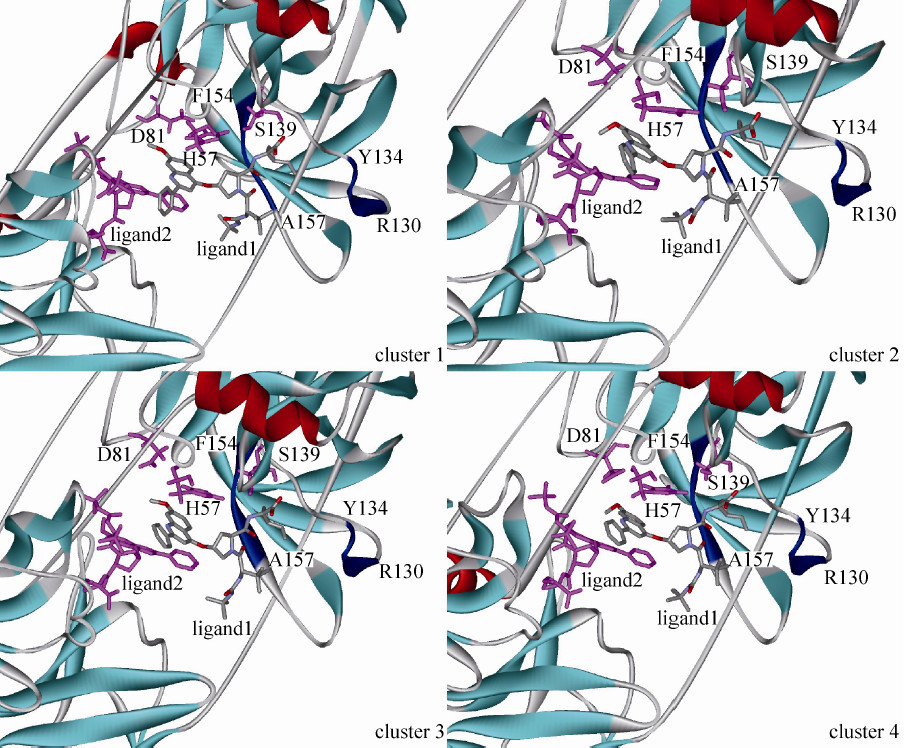
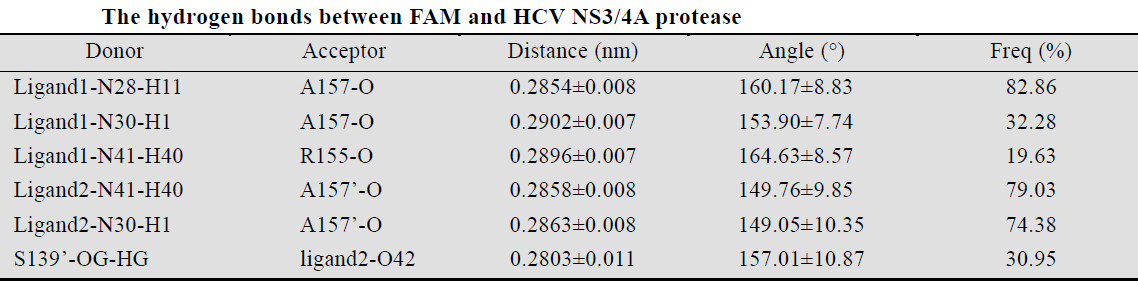
对HCV NS3/4A 蛋白酶与底物FAM 的复合物进行20.4 ns 的显含水分子动力学模拟。基于RMSF 计算值找出了分子的柔性区域,并与实验B 因子值进行了相关性分析,良好的相关性验证了模拟方法的可靠性。FAM 的取代尿素侧链上的NH 与蛋白酶A157,以及乙烯基侧链的O 与S139 形成较为稳定的氢键。
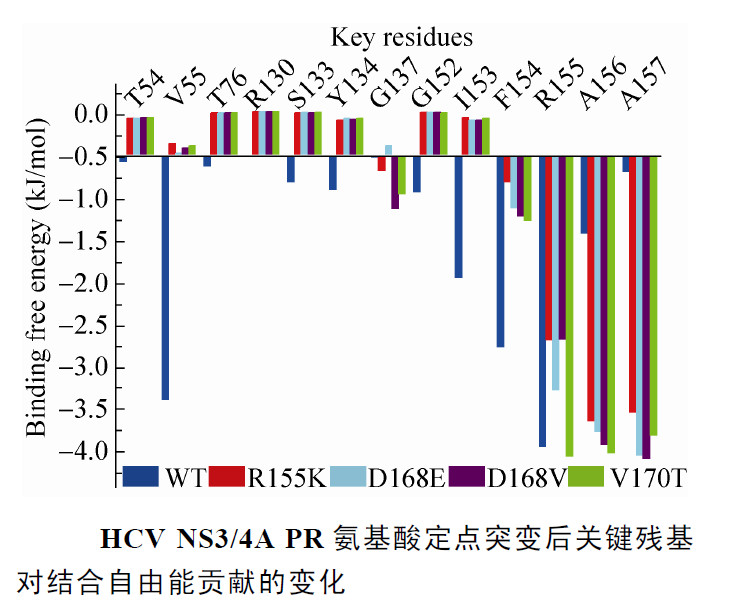
结合自由能计算值与类似化合物的实验值吻合较好,其中范德华相互作用为复合物形成的主要驱动力。通过能量分解分析发现,FAM 的双分子结合模式为发挥效果关键形式,V55 在形成复合物的关键残基,对现有实验数据做了有益补充。随后的突变实验也证明了V55、I153 和F154 对FAM 识别重要作用。
最后,对复合物体系的构象变化做了简要分析,得到了模拟过程中的4类代表性稳定构象。模拟结果为以HCV NS3/4A蛋白酶的分子设计和分子的作用机理研究提供了一定的理论指导。