项目说明
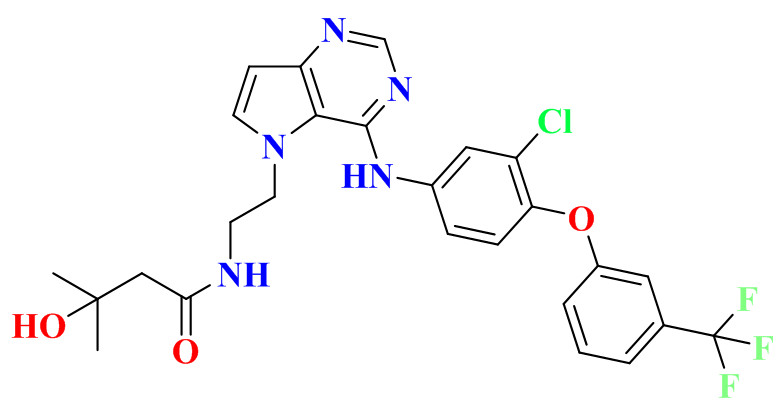
采用分子对接技术研究配体分子tak-285与EGFR激酶的结合模式。
计算方法
从RCSB Protein Data Bank(http://www.rcsb.org)下载EGFR的X-ray晶体 结构(PDB 编号:3POZ),分辨率为1.5 Å。采用UCSF Chimera 软件[1] 建立配体的三维结构,并进行能量优化。采用Dock Prep 模块添加氢原子,并分别添加AMBER ff14SB 力场和AM1-BCC 电荷[2, 3]。采用Chimera 中的DMS 工具以半径为1.4 Å 的探针生成受体的分子表面。X-ray晶体 结构显示有1 个合理的结合位点,对于该结合位点,使用sphgen 模块生成围绕活性位点的球状集合(Spheres),使用Grid 模块生成Grid 文件,该文件用于基于Grid 的能量打分评价。采用DOCK6.8 程序[4] 进行半柔性对接(semi-flexible docking),生成10000 个不同的构象取向(orientation)以及获得配体分子与结合位点的静电和范德华相互作用,并由此计算得到Grid 打分。通过聚类分析(RMSD 阈值为2.0Å),得到打分最佳的构象。最后,采用PyMOL[5]生成图片。
计算结果
结合构象打分
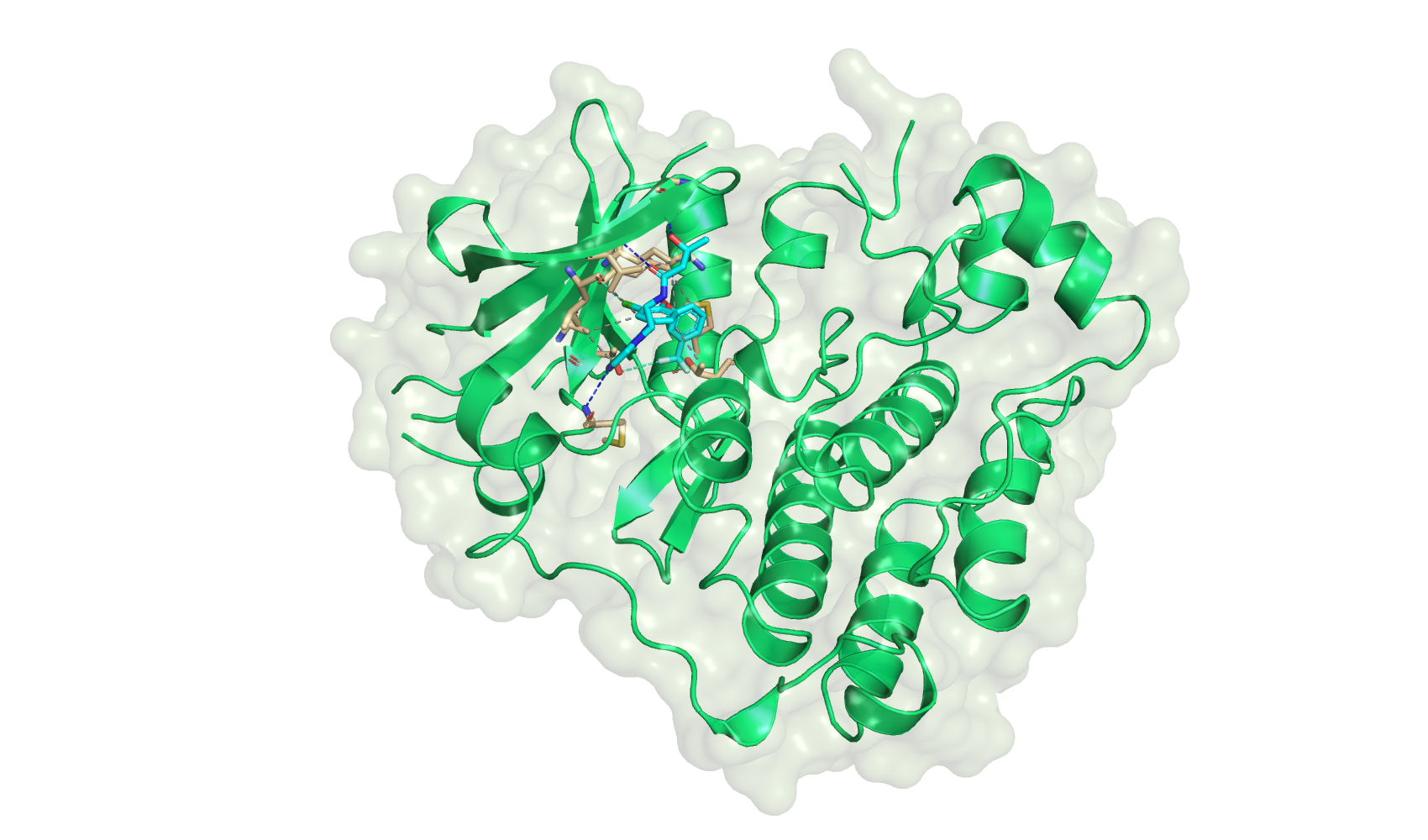
采用DOCK 6 .8程序预测tak-285 分子在EGFR中的结合模式,保留最多20 个结合构象。计算结果表明,结合位点有一个对接构象,其打分情况如下。
| Compound | Pose | Grid Score | Grid_vdw | Grid_es | Int_energy |
| tak-285 | 1 | -58.413887 | -57.743397 | -0.670492 | 6.865056 |
结合模式分析
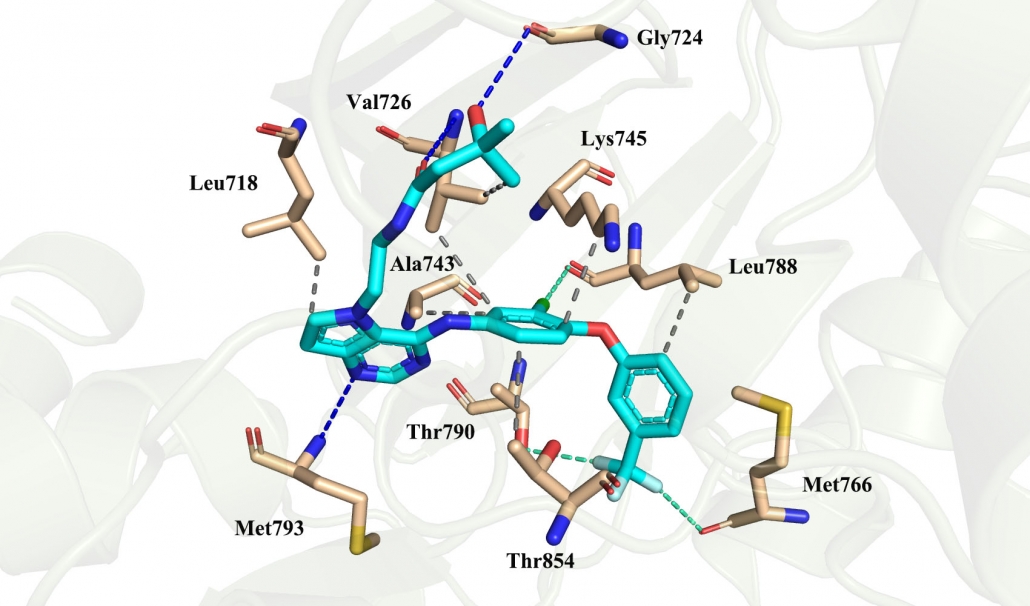
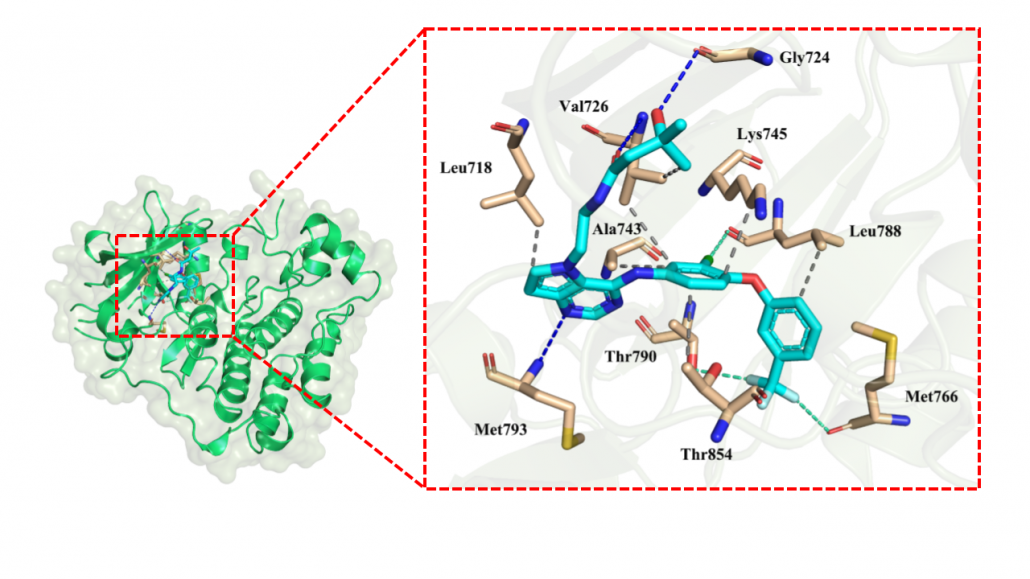
tak-285分子结构中嘧啶环上的氮原子与残基Met898形成氢键相互作用;同时,羰基氧原子和羟基氧原子与残基Val726和Gly724形成了氢键相互作用,氢键的形成使激酶与小分子之间结合更加紧密。三氟甲基上的氟原子与残基Met766和Thr790之间形成卤键,与芳环相连的氯原子与残基Leu788也形成了卤键。同时,tak-285还与残基Leu718、Val726、Ala743、Lys745、Leu788和Thr854之间形成疏水作用为分子提供了强大的范德华力。
参考文献
[1] Pettersen EF, Goddard TD, Huang CC, Couch GS, Greenblatt DM, Meng EC, and Ferrin TE.
Ucsf chimera–a visualization system for exploratory research and analysis. J Comput Chem,
25(13):1605–12, 2004.
[2] Araz Jakalian, Bruce L. Bush, David B. Jack, and Christopher I. Bayly. Fast, efficient generation
of high-quality atomic charges. am1-bcc model: I. method. Journal of Computational Chemistry,
21(2):132–146, January 2000.
[3] Araz Jakalian, David B. Jack, and Christopher I. Bayly. Fast, efficient generation of high-quality
atomic charges. am1-bcc model: I. parameterization and validation. Journal of Computational
Chemistry, 23(16):1623–1641, December 2002.
[4] Jiang, L.; Rizzo, R. C. Pharmacophore-Based Similarity Scoring for DOCK.J. Phys. Chem. B, 2015, 119 (3), 1083-1102.
[5] Schrödinger, LLC. The PyMOL molecular graphics system, version 1.8, 2015.
Support Information